| ! |
! 表示实验步骤中需要根据进行的 CUT&RUN 反应次数来调整体积的重要步骤。 |
| !! |
!! 表示需要在操作前稀释缓冲液的一个重要步骤。 |
| 安全停止 |
如果需要停止,这是实验步骤中的一个安全停止点。 |
一、细胞和组织制备
对于大多数细胞类型,活细胞可用于 CUT&RUN 检测,以产生强大的组蛋白、转录因子和辅因子富集。就某些对伴刀豆球蛋白 A 脆弱或敏感的细胞类型,略微固定细胞有助于保存细胞并保持它们完整。此外,如果使用新鲜细胞未观察到明显信号,固定可能有助于促进低丰度和/或弱结合转录因子和辅因子的富集。请注意,细胞的过度固定会抑制 CUT&RUN 检测。
我们的 CUT&RUN 分析适用于广泛的细胞或组织。如实验步骤中定义,一个 CUT&RUN 反应可包含 5,000 至 个 250,000 细胞或 1 至 5 mg 的组织。整个实验步骤中使用的缓冲液体积无需根据每次反应的细胞或组织数量进行调整,只要在此范围内即可。当需要时,缓冲液体积确实需要根据正在执行的反应数量按比例增加。如果可能,我们建议每次反应使用 100,000 个细胞或 1 mg 组织。如果细胞数量有限,我们建议每个反应至少使用 5,000 到 10,000 个细胞进行组蛋白修饰,每个反应至少使用 10,000 到 20,000 个细胞进行转录因子或辅因子。
注: 推荐用于细胞透化的毛地黄皂苷的量过多,应该足以用于大多数细胞系和组织类型的透化。然而,并非所有细胞系和组织对毛地黄皂苷都表现出相同的敏感性。如果您的特定细胞系或组织在推荐的毛地黄皂苷浓度下不起作用,您可以按照附录 A 中提供的实验步骤来优化条件。毛地黄皂苷处理应导致 >90% 的细胞群通透。
A. 活细胞制备
开始之前:
! 所有缓冲液的体积都应根据正在进行的 CUT&RUN 反应次数按比例增加。
- 在室温下收集新鲜的细胞培养物,以最大程度减少细胞应激。每个反应收集 5,000 个至 100,000 个细胞,并为 input 样品额外收集 5,000 至 100,000 个细胞。确保包括用于阳性对照 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 和阴性对照 Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362 的反应。
注: 对于贴壁细胞,首先需要使用胰蛋白酶将细胞从培养皿中分离出来,并用至少 3 体积的组织培养基中和。我们不建议从培养皿中刮取细胞,因为这会压迫甚至裂解细胞。应使用血细胞计数器或其他细胞计数器对细胞进行计数,以确保用于实验的细胞数量正确。
- 将细胞悬液在室温下以 600 x g 离心分离 3 分钟,然后移除液体。
注: 处理低细胞数(< 100,000 个总细胞)的挑战在于,离心后的细胞颗粒并不总是肉眼可见,因此很容易在洗涤步骤中丢失细胞。因此,当处理低细胞数时,我们建议跳过下面的洗涤步骤 3 到 5。伴刀豆球蛋白 A 珠与细胞的结合耐受结合反应中有 40% 的细胞培养基。因此,在步骤 2 中对细胞悬浮液进行初始离心后,可以去除大部分上清液,每次反应留下 ≤40 细胞培养基。然后在步骤 6 中,向细胞悬液中加入足够的 1X 洗涤缓冲液(+ 亚精胺 + PIC),使每次反应的总体积达到 100 µl。
- 于室温下用 1 ml 1X 洗涤缓冲液(+ 亚精胺 + PIC)中重悬细胞沉淀物,并上下轻轻吹打。
- 在室温下以 600 x g 离心分离 3 分钟,然后移除液体。
- 重复步骤 3 和 4 一次,以再次清洗细胞沉淀物。
- 对于每个反应或 input 样品,添加 100 µl 1X 洗涤缓冲液(+ 亚精胺 + PIC),并轻轻上下摇晃移液管来重悬细胞沉淀物。
- 将 100 µl 细胞转移到一个新管中,并在 4°C 下储存,直到第五部分。这是 input 样本。
注: 在随后的实验步骤中,将在 55°C 下孵育输入样品,因此建议使用一根可封盖的 1.5 ml 试管,以减少孵育过程出现蒸发。
- 立即进入第二部分。
B. 固定细胞制备
注: 固定细胞制备需要以下试剂,不包含在此试剂盒中:37% 甲醛或 16% 甲醛无甲醇 #12606、甘氨酸溶液 (10X) #7005 和 10% SDS 溶液 #20533。
开始之前:
! 所有缓冲液的体积都应根据正在进行的 CUT&RUN 反应次数按比例增加。
- 取出并温热 200X 蛋白酶抑制剂混合物 #7012 和 100X 亚精胺 #27287。确保两种物质完全融化。请注意,由于含有 DMSO,Protease Inhibitor Cocktail #7012 置于冰上时会重新冻结。
- 制备 1X 洗涤缓冲液(每种细胞系 2 ml,每个反应或每份input样品额外准备 100 µl)。例如,要制备 2.5 ml 1X 洗涤缓冲液,添加 250 µl 10X 洗涤缓冲液 #31415 + 25 µl 100X 亚精胺 #27287 + 12.5 µl 200X 蛋白酶抑制剂混合物 #7012 + 2212.5 µl Nuclease-free Water #12931。让其平衡到室温,以最大程度减少细胞应激。
- 每 1 ml 待处理细胞悬液制备 2.7 µl 37% 甲醛或 6.25 µl 16% 无甲醇的甲醛 #12606 并保存在室温下。使用制造商规定的有效期内的新鲜甲醛。
- 每个抗体/微球菌核酸酶 (MNase) 反应收集 5,000 至 100,000 个细胞,并为 input 样品额外收集 5,000 至 100,000 个细胞。确保包括用于阳性对照 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 和阴性对照 Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362 的反应。
注: 对于贴壁细胞系,首先需要使用胰蛋白酶将细胞从培养皿中分离出来,并用至少 3 体积的培养基中和。我们不建议从培养皿中刮取细胞,因为这会压迫甚至裂解细胞。应使用血细胞计数器或其他细胞计数器对细胞进行计数,以确保用于实验的细胞数量正确。
- 每 1 ml 细胞悬液添加 2.7 µl 37% 甲醛或 6.25 µl 16% 无甲醇的甲醛 #12606,以达到 0.1% 甲醛的最终浓度。旋转混匀并置于室温下孵育 2 分钟。
- 每 1 ml 固定细胞悬液添加 100 µl 甘氨酸溶液 (10X) #7005 以停止交联。旋转混匀并置于室温下孵育 5 分钟。
- 将细胞悬液在 4°C 下以 3,000 x g 的速度离心 3 分钟并除去液体。立即进入第 5 步。(安全停止)或者,固定的细胞沉淀可以在使用前储存在 -80°C 下长达 6 个月。
注: 处理低细胞数(< 100,000 个总细胞)的挑战在于,离心后的细胞颗粒并不总是肉眼可见,因此很容易在洗涤步骤中丢失细胞。在这种情况下,我们不建议冷冻细胞沉淀物。此外,当处理这些低细胞数时,我们建议跳过下面的洗涤步骤 5 到 7。伴刀豆球蛋白 A 珠与细胞的结合耐受结合反应中有 40% 的细胞培养基。因此,在步骤 4 中对细胞悬浮液进行初始离心后,可以去除大部分上清液,每次反应留下 ≤40 细胞培养基。然后在步骤 8 中,向细胞悬液中加入足够的 1X 洗涤缓冲液(+ 亚精胺 + PIC),使每次反应的总体积达到 100 µl。
- 通过上下轻轻吹打,在 1 ml 1X 洗涤缓冲液(+ 亚精胺 + PIC)中重悬细胞沉淀物。
- 在室温下以 3,000 x g 的速度离心分离 3 分钟,然后移除液体。
- 重复步骤 5 和 6 一次,以再次清洗细胞沉淀物。
- 对于每个反应或 input 样品,添加 100 µl 1X 洗涤缓冲液(+ 亚精胺 + PIC),并轻轻上下摇晃移液管来重悬细胞沉淀物。
- 将 100 µl 细胞转移到一个新管中,并在 4°C 下储存,直到第五部分。这是 input 样本。
注: 在随后的实验步骤中,将在 55°C 下孵育输入样品,因此建议使用一根可封盖的 1.5 ml 试管,以减少孵育过程出现蒸发。
- 立即进入第二部分。
C. 组织样品制备
对于大多数组织类型,1 mg 轻微固定的组织(0.1% 甲醛,2 分钟)可以产生强大的组蛋白、转录因子和辅因子富集。富集组蛋白修饰不需要甲醛固定。然而,许多转录因子和辅因子确实需要对组织进行光固定以获得最佳结果。一些低丰度和/或弱结合转录因子和辅助因子可能需要中等固定(0.1% 甲醛,10 分钟)以获得最佳结果。此外,在使用具有挑战的组织类型(如纤维组织)时,中等固定可能会改善结果。请注意,过度固定会抑制 CUT&RUN 检测。固定的组织样本在使用前可以在 -80°C 下冷冻长达 6 个月。
注: 为 CUT&RUN 准备新鲜组织(无固定)时,我们建议在制备组织之前制备伴刀豆球蛋白 A 珠(第 II 部分,步骤 1 到 5),以尽量减少细胞在珠子制备过程中停留的时间。活化的珠子可以储存在冰上直到准备使用。
注: 固定组织制备需要以下试剂,不包括在此试剂盒中:37% 甲醛或 16% 甲醛无甲醇 #12606、磷酸盐缓冲盐水 (PBS) #9872、甘氨酸溶液 (10X) #7005 和 10% SDS 溶液 #20533。
开始之前:
! 所有缓冲液的体积都应根据正在进行的 CUT&RUN 反应次数按比例增加。
- 取出并温热 200X 蛋白酶抑制剂混合物 #7012 和 100X 亚精胺 #27287。确保两种物质完全融化。请注意,由于含有 DMSO,Protease Inhibitor Cocktail #7012 置于冰上时会重新冻结。
- 制备 1X 洗涤缓冲液(每种组织类型 3 ml,每个反应或每份 input 样品额外准备 100 µl)。例如,要制备 3.5 ml 1X 洗涤缓冲液,添加 350 µl 10X 洗涤缓冲液 #31415 + 35 µl 100X 亚精胺 #27287 + 17.5 µl 200X 蛋白酶抑制剂混合物 #7012 + 3097.5 µl Nuclease-free Water #12931。让其平衡到室温,以最大程度减少细胞应激。
- 如果需要组织固定,请准备以下缓冲液:
- 为每种组织类型制备 1 ml 固定缓冲液,方法是通过添加 2.7 µl 37% 甲醛或 6.25 µl 16% 无甲醇的甲醛 #12606 和 5 µl 200X 蛋白酶抑制剂混合物 (PIC) #7012 到 1 ml 磷酸盐缓冲盐水 (PBS) #9872 中。使用制造商规定的有效期内的新鲜甲醛。
- 为每种组织类型准备 1 ml PBS #9872 + 5 µl 蛋白酶抑制剂混合物 (PIC) #7012 并置于冰上。
- 每 1 ml 固定缓冲液制备 100 µl 甘氨酸溶液 (10X) #7005。
- 为每个抗体/MNase 反应称量 1 mg 新鲜组织,并为 input 样本称量额外的 1 毫克组织。确保包括用于阳性对照 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 和阴性对照 Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362 的反应。
注: 对于某些转录因子或辅因子,或对于诸如纤维组织之类的困难组织类型,每个反应最多可使用 5 mg 组织而无需按比例放大试剂。
- 将组织样本放入盘中,并使用干净的手术刀或剃须刀片切碎。将培养皿放在冰上。重要的是将组织置于低温下以防止蛋白质降解。
注: 我们建议对组织进行光固定,因为这种条件最适合大多数组织类型和蛋白质靶标。但是,如果需要新鲜组织,请跳过步骤 3 到 8 并立即进行步骤 9。
- 立即将切碎的组织转移到 1 ml 的固定溶液中,并旋转试管以混匀。
注: 此体积的固定溶液足以容纳多达 50 mg 的组织。如果处理 >50 mg,则在步骤 7 中相应地增加固定溶液和 1X PBS + PIC 溶液。
- 室温孵育 2 分钟。
注: 对于困难的组织类型(如纤维组织)或低丰度和/或弱结合转录因子或辅因子,将甲醛固定延长至 10 分钟可能会改善结果。
- 通过每 1 ml 固定缓冲液添加 100 µl 甘氨酸溶液 (10X) #7005 来停止交联。旋转混匀并置于室温下孵育 5 分钟。
- 在室温下以 2,000 x g 的速度离心分离 5 分钟,然后移除液体。
- 用 1 ml 1X PBS + PIC 重悬组织。
- 在 4°C 下以2,000 x g 的速度离心 5分钟,然后移除液体,继续步骤 9。(安全停止) 或者,固定的组织沉淀物可以在解聚前在 -80°C 下储存长达 6 个月。
- 将组织重悬在 1 ml 1X 洗涤缓冲液(+ 亚精胺 + PIC)中,并将样品转移到 Dounce 匀浆器中。
- 用 20-25 冲程将组织块分解成单细胞悬浮液,直到观察不到组织块。
- 将细胞悬液转移到 1.5 ml 管中,并在室温下以 3,000 x g 的速度离心 3 分钟,从细胞中去除上清液。
- 在 1 ml 1X 洗涤缓冲液(+ 亚精胺 + PIC)中重悬细胞沉淀。
- 将细胞悬液在室温下以 3,000 x g 离心分离 3 分钟,然后移除液体。
- 重复步骤 12 和 13 一次,以再次清洗细胞沉淀物。
- 针对每个反应,添加 100 µl 1X 洗涤缓冲液(+ 亚精胺 + PIC),并轻轻上下摇晃移液管来重悬细胞沉淀物。
- 将 100 µl 细胞转移到一个新管中,并在 4°C 下储存,直到第五部分。这是 input 样本。
注: 在随后的实验步骤中,将在 55°C 下孵育输入样品,因此建议使用一根可封盖的 1.5 ml 试管,以减少孵育过程出现蒸发。
- 立即进入第二部分。
II. 刀豆球蛋白 A 珠子和一抗的结合
开始之前:
! 所有缓冲液的体积都应根据正在进行的 CUT&RUN 反应次数按比例增加。
- 通过轻轻上下吹打小心地重悬 Concanavalin A 磁珠,确保不要将任何磁珠悬浮液溅出试管。按照每个 CUT&RUN 反应,将 10 µl 珠子悬液转移到一个新的 1.5 ml 微量离心管中。
注: 避免涡旋伴刀豆球蛋白 A 珠悬浮液,因为反复涡旋可能会从磁珠中置换出伴刀豆球蛋白 A。
- 每 10 µl 珠子添加 100 µl Concanavalin A Bead Activation Buffer。上下吸打来轻轻混合珠子。
- 将试管放在磁力架上,直到溶液变澄清(30 秒至 2 分钟),然后移除液体。
注: 为了避免磁珠丢失,请使用移液枪来移除液体。请勿使用真空设备抽吸。
- 从磁力架上取下试管。重复步骤 2 和 3 一次,再次清洗珠子。
- 添加一体积的 Concanavalin A Bead Activation Buffer,体积等同于初始的重悬珠子的液体体积(每份样品 10 µl),上下吹打重悬。
注: 如果在细胞或组织制备之前制备伴刀豆球蛋白 A 珠(如建议用于活细胞和新鲜组织),则活化的珠子可以储存在冰上直至使用。
- 确保将伴刀豆球蛋白 A 珠充分混合到溶液中。在 I-A 部分步骤 8、I-B 部分步骤 10 或 I-C 部分步骤 17 中制备的洗涤细胞悬浮液中,每次反应添加 10 µl 活化珠悬浮液。
- 通过上下移液将样品充分混合。室温孵育 5 分钟。
注: Concanavalin A 磁珠可能会结块或粘附在试管壁上。上下吹打来重悬珠子。无需摇动或振摇样品样管。
- 将试管放在磁力架上,直到溶液变澄清(30 秒钟至 2 分钟),然后取下试管并丢弃液体。
- 从支架上取下试管。添加 100 µl 抗体结合缓冲液(+ 亚精胺 + PIC + 洋地黄皂苷),并放在冰上。
- 按每次反应分装 100 µl 细胞珠子悬液到一根单独的 1.5 ml 试管中,并放在冰上。
- 按每次反应添加适量抗体,并上下吹打来轻柔混合。
注: CUT&RUN 需要的抗体量会有所不同,应由使用者确定。对于阳性对照 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751,向样品中添加 2 µl 抗体。对于阴性对照 Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362,向样品中添加 5 µl。我们强烈建议使用阴性对照抗体,而不是非抗体对照,因为后者会导致高水平的非特异性 MNase 消化和高背景信号。我们建议尽可能使用 input 样本与 qPCR 和 NG-seq 分析进行比较。
- 在 4°C 下孵育 2 小时。该步骤可以延长到过夜。
注: Concanavalin A 磁珠可能会结块或粘附在试管壁上。上下吹打来重悬珠子。无需摇动或振摇样品样管。
III. pAG-MNase 酶结合
开始之前:
! 所有缓冲液的体积都应根据正在进行的 CUT&RUN 反应次数按比例增加。
- 来自第 II 部分,步骤 12 的管置于磁力架上,直到溶液变澄清(30 秒至 2 分钟),然后移除液体。
- 从磁力架上取下试管,添加 1 ml 洋地黄皂苷缓冲液(+ 亚精胺 + PIC + 洋地黄皂苷)。通过轻轻上下移液重悬珠子,确保收集粘在管壁上的所有珠子。
- 将试管放在磁力架上,直到溶液变澄清(30 秒钟至 2 分钟),然后移除液体。
- 从磁力架上取下试管。向每根试管添加 50 µl pAG-MNase 预混合物,并轻轻上下吹打来混合样品。
- 在 4°C 下孵育 1 小时。
注:Concanavalin A 磁珠可能会结块或粘附在试管壁上。上下吹打来重悬珠子。无需摇动或振摇样品样管。
- 立即进行第四部分。
IV. DNA 消化和释放
开始之前:
! 所有缓冲液的体积都应根据正在进行的 CUT&RUN 反应次数按比例增加。
- 取出洋地黄皂苷溶液 #16359 并将其加热至 90-100°C 达 5 分钟,并确保其完全解冻并处于溶液中。立即将已融化的洋地黄皂苷溶液 #16359 放在冰上。
注: 洋地黄皂苷溶液 #16359 应保存在 -20°C 下。请在使用过程中将其保存在冰上,并在实验完成后保存在 -20°C 下。
- 将氯化钙放在冰上。
- 如果从第 I 节中的固定材料开始,请确保 10% SDS Solution #20533 完全溶解。加热至 37°C 将有助于溶解 SDS 沉淀物。
- 每次反应制备 150 µl 1X Stop Buffer(37.5 µl Stop Buffer #48105 + 3.75 µl 洋地黄皂苷溶液 #16359 + 0.75 µl 200X RNAse A #7013 + 108 µl Nuclease-free Water #12931)。
可选:如需对样品进行标准校对,则可将 Sample Normalization Spike-In DNA 添加到 1X Stop Buffer 中(例如,见第 VII 部分的图 8)。对于 qPCR 分析,我们建议每次反应加入 5 µl (5 ng) Spike-In DNA。对于 NG-seq 分析,我们建议用 Nuclease-free Water #12931 稀释 Sample Normalization Spike-In DNA 100 倍,每次反应加入 5 µl (50 pg) Spike-In DNA。当每次反应使用 100,000 个细胞或 1 mg 组织时,这可确保标准化读数约为总测序读数的 0.5%。如果每个反应使用多于或少于 100,000 个的细胞或 1 mg 组织,按比例放大或缩小样本标准化 Spike-In DNA 的体积,以将标准化读数调整到总读数的 0.5% 左右。
- 来自第 III 部分,步骤 6 的管置于磁力架上,直到溶液变澄清(30 秒至 2 分钟),然后移除液体。
- 从磁分离架上取下试管。添加在第 III 部分配制的 1 ml 毛地黄皂苷缓冲液(+ 亚精胺 + PIC + 毛地黄皂苷),并通过柔和地上下吹打来重悬珠子。
- 重复步骤 2 和 3 一次。
- 将试管放在磁力架上,直到溶液变澄清(30 秒钟至 2 分钟),然后移除液体。
- 从磁力架上取下试管。添加在第 III 部分配制的 150 µl 毛地黄皂苷缓冲液(+ 亚精胺 + PIC + 毛地黄皂苷)至每个管,并通过上下吹打来混合。
- 将试管在冰上放置 5 分钟,使之在消化反应前冷却。
- 向每根试管中添加 3 µl 冷却的氯化钙来激活 pAG-MNase,并上下吹打混合。
- 4°C 条件下孵育样本 30 分钟。
注: 应在 4°C 的温控板上或在冰箱中进行消化。冰的温度可低至 0°C,这可能会限制消化并降低信号。无需摇动或振摇样品样管。
- 向每份样品中添加 150 µl 1X Stop Buffer(+ 洋地黄皂苷 + RNAse A + Spike-in DNA [可选]),并上下吹打来混合。
- 在 37°C 下孵育试管 10 分钟,不要摇晃,使 DNA 片段进入溶液。
- 以 16,000 x g 在 4°C 下离心分离 2 分钟,然后将试管放在磁力架上,直到溶液变澄清(30 秒钟至 2 分钟)。
- 将上清液转移到一根新的 2 ml 微量离心管中。 这些是富含染色质的样品。
注: 如果使用活细胞或新鲜组织(未固定)进行 CUT&RUN 测定,跳过步骤 13-14,然后立即进行步骤 15。
注: 在随后的实验步骤中,将在 65°C 下孵育固定样品,因此建议使用一根可封盖的2 ml 试管,以减少孵育过程出现蒸发。
- 要逆转固定细胞或组织样品中的交联,让样品升温至室温并添加 3 µl 10% SDS 溶液 #20533(0.1% 最终浓度)和 2 µl 蛋白酶 K (20 mg/mL) #10012 到每个样品。
注: 如果样品没有预热到室温,SDS 可能会从溶液中沉淀出来。
- 涡旋混合每个样品并在 65℃ 下孵育样品 2 小时。 这次孵育可以延长过夜。孵育后,以 10,000 x g 的速度快速旋转样品 1 秒,以收集管盖的蒸发。
- 将样品平衡至室温并进行第 VI 部分。(安全停止)或者,可将样品保存在 -20°C 下, 1 周以内。但在 DNA 纯化(第 VI 部分)之前,请确保将样品加热到室温。
V. Input 样品的制备
输入 DNA 的片段化是与下游 NG 测序兼容所必需的,但对于下游 qPCR 分析则不是必需的。如果没有超声波仪,我们建议使用未片段化的输入 DNA 进行 qPCR 分析;然而,输入 DNA 应使用苯酚/氯仿提取和乙醇沉淀进行纯化,因为未片段化输入 DNA 的尺寸太大而无法使用 DNA 离心柱进行纯化。如果没有超声波仪并且需要进行下游 NG 测序分析,则可以使用 CUT&RUN 正常 IgG 抗体样品作为阴性对照,尽管这并不理想,因为正常的富含 IgG 的样品可能会显示非特异性 DNA 富集。或者,使用 MNase 的输入 DNA 片段化实验步骤可在 https://cst-science.com/CUT-RUN-input-digestion 获得。
! 所有缓冲液的体积都应根据正在制备的input样品的数量按比例增加。
开始之前:
- 取出并加热 DNA 提取缓冲液 #42015。确保完全解冻。
- 每份 input 样品制备 2 µl 蛋白酶 K #10012 + 0.5 µl RNAse A #7013 + 197.5 µl DNA 的提取缓冲液 #42015(每份 input 样品共 200 µl)。
- 将 200 µl DNA 提取缓冲液(+ 蛋白酶 K + RNAse A)添加到 100 µl input 样品中,这些样品来自 I-A 部分步骤 7、I-B 部分步骤 9 或 I-C 部分步骤 16。上下吹打混合。
- 将试管放在 55°C 下 1 小时,伴有摇晃。
- 将试管放在冰上 5 分钟,让样品完全冷却。
- 对input样品进行超声处理,以裂解细胞并破碎染色质。在两次脉冲之间将样品放在冰上孵育 30 秒钟。
注: 超声处理条件可能需要根据附录 B 中的协议通过测试不同的超声仪功率设置和/或超声处理持续时间来凭经验确定。最佳超声处理条件将产生大小从 100-600 bp 不等的染色质片段。使用设置为 6 且配有 1/8 英寸探针的 VirTis Virsonic 100 超声波均质器/超声波仪以 5 组 15 秒脉冲进行超声处理,可充分碎裂input染色质。
- 在 4°C 下以 18,500 x g 离心分离 10 分钟来使裂解物变澄清。将上清液转移到一根新的 2 ml 微量离心管。
- 立即进行第六部分“DNA 纯化”。(安全停止)或者,可将样品保存在 -20°C 下, 1 周以内。但在 DNA 纯化程序(第 VI 部分)之前,确保将样品加热到室温。
VI. DNA 纯化
如 VI - A 部分所述,使用 DNA 离心柱可从input和富集染色质样品中纯化 DNA,或如第 VI - B 部分所述,使用苯酚/氯仿提取后再行乙醇沉淀方法进行纯化。使用 DNA 离心柱纯化简单快速,可很好地回收 35 bp 以上的 DNA 片段(图 7A,泳道 2)。苯酚/氯仿提取之后再行乙醇沉淀更加困难,但能很好地回收 35 bp 以下的 DNA 片段(图 7A,泳道 3);但如图 7B 所示,大多数在 CUT&RUN 检测中产生的 DNA 片段大于 35 bp。因此,DNA 离心柱提供一种快速简单的方法来纯化 > 98% 的所有 CUT&RUN DNA 片段。
在 NG-seq 分析之前,使用基于 picogreen 的 DNA 定量测定法可以对纯化的 DNA 进行定量。对于含 100,000 个细胞的 CUT&RUN 反应,一次转录因子和辅因子的 CUT&RUN 反应中,预期 DNA 产量为 0.5-10 ng,而在一次组蛋白修饰反应中则为 1-20 ng。
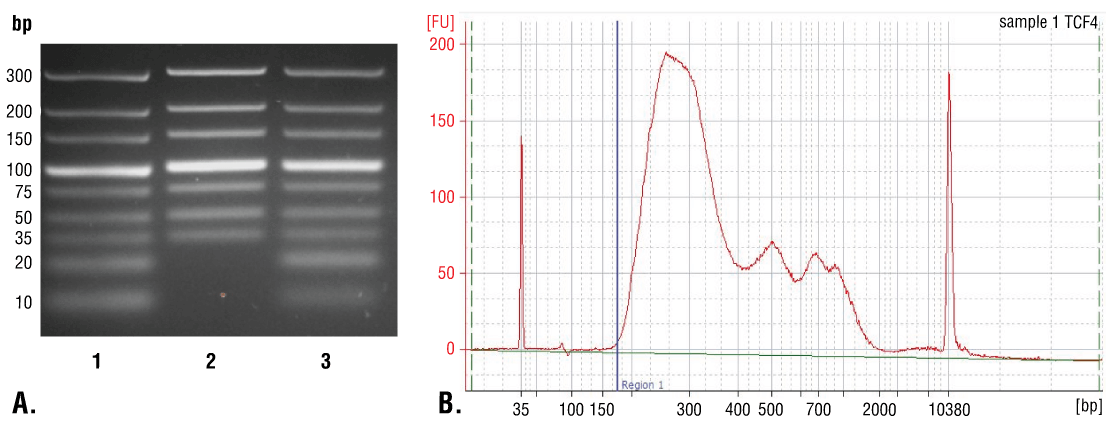
图 7. 比较使用离心柱或苯酚/氯仿提取后再行乙醇沉淀方法进行的 DNA 纯化。(A) 将低量程 DNA 标准品混合物(泳道 1,未纯化)使用 DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN,CUT&Tag) #14209(泳道 2)或苯酚/氯仿提取后接乙醇沉淀法(泳道 3)纯化,并通过在 4% 琼脂糖凝胶上电泳来分离。如图所示,苯酚/氯仿提取后再行乙醇沉淀能有效回收所有大小的 DNA 片段,而 DNA 离心柱仅能回收 ≥ 35 bp 的 DNA 片段。(B) 在使用 TCF4/TCF7L2 (C48H11) Rabbit mAb #2569 的 CUT&RUN 检测中使用苯酚/氯仿提取后再行乙醇沉淀方法来纯化 DNA。使用一台Bioanalyzer (Agilent Technologies) 分析文库中 DNA 片段的大小。构建期间向文库添加的接头和条码序列占了 140 bp 片段长度。因此,文库制备后,起始 35 bp 的 DNA 片段长度将变为 175 bp(图中用蓝色垂直线标注)。如图所示,总 CUT&RUN 富集 DNA 片段中不到 2% 短于 175 bp(起始长度 35 bp),提示 DNA 纯化离心柱足以捕获 > 98% 的总 CUT&RUN DNA 片段。
A. 使用离心柱进行 DNA 纯化
注: 使用 DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN, CUT&Tag) #14209(未在本试剂盒中包含)以及下面改良的实验步骤,可以从输入的和富集的染色质样品中纯化 DNA。步骤 1-5 已修改,以符合向 300 µl input样品和富集染色质样品中添加 5 倍体积 (1.5 ml) DNA 结合缓冲液的要求。
在开始之前:
- !! 使用前添加 24 ml 乙醇 (96-100%) 到 DNA Wash Buffer 中。此操作仅需在第一次DNA纯化前进行。
- 针对每份需纯化的富集染色质样品或 input 样品,取出一个 DNA 纯化收集管。
- 向每份 input 样品和富集染色质样品中添加 1.5 ml DNA 结合缓冲液,并短暂涡旋。
注: 对于每 1 体积样品,应当使用 5 倍体积的 DNA 结合缓冲液。
- 从在步骤 1 中制备的每份样品中取出 600 μl 并将其至收集管的 DNA 离心柱中。
- 用离心机以 18,500 x g 离心 30 秒。
- 从收集管取出离心柱并丢弃液体。将离心柱放回空的收集管。
- 重复步骤 2-4,直到步骤 1 中的整个样品已经过离心柱旋转。将离心柱放回空的收集管。
- 向收集管的离心柱添加 750 μl DNA Wash Buffer。
- 用离心机以 18,500 x g 离心 30 秒。
- 从收集管取出离心柱并丢弃液体。将离心柱放回空的收集管。
- 用离心机以 18,500 x g 离心 30 秒。
- 丢弃收集管和液体。保留离心柱。
- 向每根离心柱添加 50 μl DNA 洗脱缓冲液,并放在干净的 1.5 ml 管中。
- 用微量离心机以 18,500 x g 的离心力离心分离 30 秒,以洗脱 DNA。
- 取出并丢弃 DNA 离心柱。洗脱物即纯化的 DNA。(安全停止)样本在 -20°C 下最多保存 6 个月。
B. 使用苯酚/氯仿提取和乙醇沉淀方法进行 DNA 纯化
注: 苯酚/氯仿萃取和乙醇沉淀需要以下试剂,但不包含在该试剂盒中:苯酚/氯仿/异戊醇(25:24:1)、氯仿/异戊醇 (24:1)、3M 醋酸钠 (pH 5.2)、20mg/mL 糖原、100% 的乙醇、70% 的乙醇和 1X TE 缓冲液或 Nuclease-free Water #12931。
- 向每份input样品和富集染色质样品中添加 300 µl 苯酚/氯仿/异戊醇 (25:24:1),并涡旋 30 秒钟充分混合。
- 使用微量离心机以 16,000 x g 的离心力离心 5 分钟来分层。小心地将大部分顶部水层(避开中间层)转移到一个新管中。
- 向液体样品中添加 300 µl 氯仿/异戊醇 (24:1),并涡旋 30 秒钟充分混合。
- 使用微量离心机以 16,000 x g 的离心力离心 5 分钟来分层。小心地将大部分顶部水层(避开中间层)转移到一个新管中。
- 向每份液体样品中添加 25 µl 3M 乙酸钠 (pH 5.2)、1 µl 20mg/mL 糖原和 600 µl 100% 乙醇,并涡旋 30 秒钟混合。
- 样品在 -80°C 下孵育 1 小时或在 -20°C 下孵育过夜来使 DNA 沉淀。
- 用微量离心机在 4°C 下以 16,000 x g 离心 5 分钟来使 DNA 沉淀。
- 小心地移除上清液,用 70% 乙醇洗涤沉淀物。
- 用微量离心机在 4°C 下以 16,000 x g 离心 5 分钟来使 DNA 沉淀。
- 移除上清液,并风干沉淀物。
- 在 50 µl 1X TE 缓冲液或无核酸酶水 #12931 中重悬沉淀物。这是纯化的 DNA。(安全停止)样本在 -20°C 下最多保存 6 个月。
VII. 用 qPCR 定量 DNA
建议:
- 试剂盒中包含的Sample Normalization Primer Set 针对酿酒酵母 ACT1 基因,可用于定量Sample Normalization Spike-In酵母 DNA 的信号来进行样品标准化(可选)。
- 试剂盒中包含的额外对照引物专用于人或小鼠 RPL30 基因(#7014 or #7015),可用来对 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 样品进行定量实时 PCR 分析。如果用户对其他物种进行 CUT&RUN,则用户需要为该物种设计相应的对照引物,并确定最佳 PCR 条件。
- PCR 引物选择至关重要。对于 CUT&RUN,PCR 扩增子大小应大约为 60-80 bp 长。引物的最佳熔解温度应设计为 60°C 左右,GC 含量应在 50% 左右。
- 2 µl 纯化的 DNA 足以对组蛋白、转录因子和辅因子的靶基因进行 qPCR 介导的定量。
- 建议使用热启动 Taq 聚合酶以最大限度降低非特异性 PCR 产物的风险。
- 使用带滤嘴的移液器吸头,从而最大限度地减少污染风险。
- 标记适当数量、兼容待用 PCR 仪型号的 PCR 管或 PCR 平板。PCR 反应应当包括阳性对照三甲基组蛋白 H3 Lys4 样品、阴性对照兔 IgG 样品、无 DNA 的管(作为 DNA 污染的对照)以及 DNA 输入样品。如果需要,可以使用输入 DNA 的系列稀释物(未稀释 - 100% 输入,1:5 - 20% 输入,1:25 - 4% 输入,1:125 - 0.8% 输入)创建标准曲线并确定扩增效率及定量每个免疫富集的样品中 DNA 的量。
注:如果进行样品标准化,仅 CUT&RUN 样品使用 Sample Normalization Primer Set 分析 。input DNA 不包含Normalization Spike-In DNA。
- 向 PCR 平板上的每只管或每个孔中添加 2 μl 相应的 DNA 样品。
- 按下文所述的步骤制备主反应混合物。每次 PCR 反应设置 2-3 次复孔。添加足量试剂来弥补损耗的体积(1-2 次额外反应)。向每支 PCR 反应管或每个孔中添加 18 μl 反应混合物。
| 试剂 |
1 次 PCR 反应所需体积 (18 μl) |
| Nuclease-free H2O #12931 |
6 μl |
| 5 µM 引物 |
2 μl |
| SimpleChIP® Universal qPCR Master Mix #88989 |
10 μl |
- 启动以下 PCR 反应程序:
| a. |
初始变性 |
95°C,3 分钟 |
| b. |
变性 |
95°C,15 秒钟 |
| c. |
退火和延伸 |
60°C,60 秒钟 |
| d. |
重复步骤 b 和 c,共循环 40 次。 |
|
- 使用实时 PCR 仪的自带软件,分析定量 PCR 结果。或者,可以使用input样品百分数方法和以下所示的公式,手动计算 IP 效率。采用这种方法,从每个抗体反应获得的信号表述为占总输入染色质的百分比。如果使用 DNA 输入样品的系列稀释物,则绘制并利用标准曲线比照输入百分比 (100%、20%、4%、0.8%) 的 Log(10) 计算从每个抗体反应所获得的信号。
- 输入百分比 = 100% x 2(C[T] 100% 输入样品 – C[T] IP 样品)
- C[T] = CT = PCR 反应的平均循环阈值
- 对于样品标准化,选择样品标准化引物组 C[T] 值最低的样品作为选定样品(例如下表示例中的样品 1),并使用以下方程式计算其他样品的标准化系数。使用各自标准化系数调整待测引物组的信号。
qPCR 检测样品标准化示例(见图 8)
| |
样品标准化引物组的 C[T] 值 |
**qPCR 的标准化系数 |
标准化之前的信号(步骤 5 中计算出的输入百分比) |
标准化之后的信号 |
| 样品 1 |
23.31 |
2(23.31-23.31)=1.00 |
24.4% |
24.4%/1.00=24.4% |
| 样品 2 |
24.24 |
2(23.31-24.24)=0.52 |
12.0% |
12.0%/0.52=23.1% |
| 样品 3 |
25.08 |
2(23.31-25.08)=0.29 |
6.28% |
6.28%/0.29=21.7% |
| 样品 4 |
26.30 |
2(23.31-26.30)=0.13 |
2.72% |
2.72%/0.13=20.9% |
**qPCR 的标准化系数 = 2(C[T] 选定样品 – C[T] 其他样品)
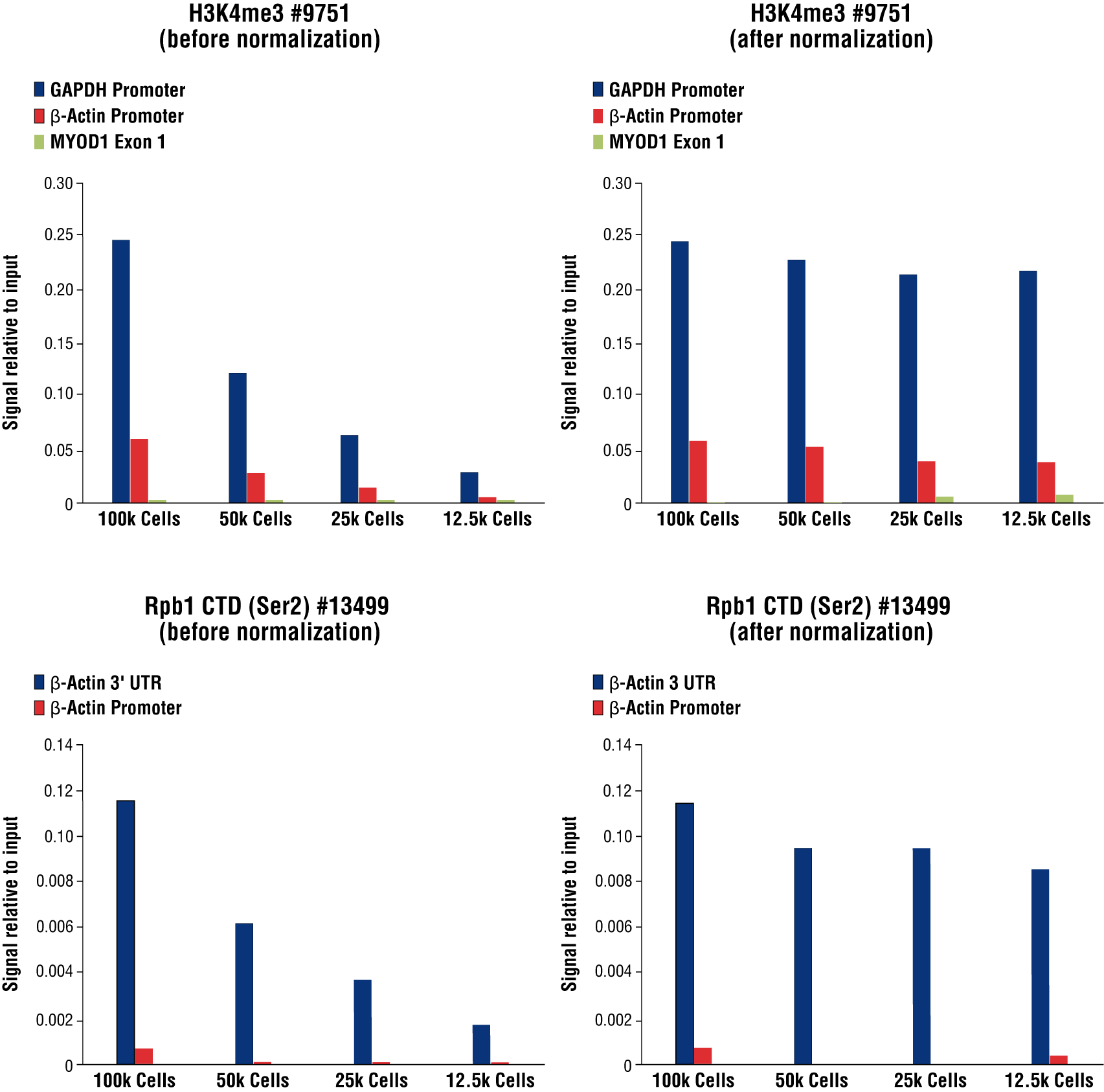
图 8. 应用添加的Spike-In DNA对 CUT&RUN 信号进行 qPCR 标准化分析。对数量渐减的 HCT116 细胞和 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 (上小图)或 Phospho-Rpb1 CTD (Ser2) (E1Z3G) Rabbit mAb #13499(下小图)进行 CUT&RUN 检测。使用 SimpleChIP® Human GAPDH Exon 1 Primers #5516、SimpleChIP® Human β-Actin Promoter Primers #13653、SimpleChIP® Human Β-Actin 3' UTR Primers #13669 和 SimpleChIP® Human MyoD1 Exon 1 Primers #4490 进行实时 PCR 来对富集的 DNA 进行定量。 每份样品中免疫沉淀 DNA 的数量表示为与 100,000 个细胞的input染色质总量相对应的信号。左图中为非标准化的富集结果。在每次反应中,按照与起始细胞数量成比例的方式添加 Sample Normalization Spike-In DNA。根据每份样品中添加 DNA 所产生的 qPCR 信号差异,将 CUT&RUN 信号标准化为含 100,000 个细胞的样品。右图显示了标准化后的富集。
VIII. NG-Sequencing文库构建
用这种试剂盒制备的免疫富集 DNA 样品直接兼容 NG-seq。要构建下游 NG-seq DNA 文库,请使用与您的下游测序平台兼容的 DNA 文库制备实验步骤或试剂盒。对于在 Illumina® 平台上测序,我们建议按照 CUT&RUN DNA 的实验步骤,使用 DNA Library Prep Kit for Illumina® (ChIP-seq, CUT&RUN) #56795 和 Multiplex Oligos for Illumina® (ChIP-seq, CUT&RUN) #29580 或 #47538 进行测序。
- CUT&RUN 中产生的背景信号很低,因此每份样品 5 百万个读数的测序深度对于组蛋白修饰和转录因子而言通常已经足够了。如果每份样品的测序深度大于 1500 万,则读取的重复率显著增加。如果每份样品的测序深度低于两百万,则信噪比会降低。
- 对于少于 20,000 个起始细胞数,在 NGS 读取中获得较低的映射率或较高的重复率是很常见的。如果发生这种情况,我们建议增加测序深度以获得足够数量的唯一映射读取用于下游数据分析。
- 进行样品标准化时,将所有样品的 CUT&RUN 测序数据比对至测试参照基因组(例如人)和样品标准化酿酒酵母基因组。选择酵母unique reads数最少的样品作为选定样品(例如下表中的样品 1),并使用下方的方程式计算其他样品的标准化系数。使用各自标准化系数来减低与每份样品的测试参考基因组对应的unique reads数。使用缩小的数据集进行进一步的 NGS 分析。
用于 NGS 分析的样本标准化示例
| |
与酵母对应的unique reads数 |
NGS 的标准化系数 |
标准化之前与测试参考基因组对应的unique reads数 |
NGS 的标准化系数 = 选定样品的酵母 reads数/其他样品的酵母unique reads数 |
| 样品 1 |
219,275 |
219,275/219,275 = 1.00 |
5,077,747 |
5,077,747 X 1.00 = 5,077,747 |
| 样品 2 |
411,915 |
219,275/411,915 = 0.53 |
9,896,671 |
9,896,671 X 0.53 = 5,268,306 |
| 样品 3 |
816,235 |
219,275/816,235 = 0.27 |
17,842,773 |
17,842,773 X 0.27 = 4,793,320 |
| 样品 4 |
1,120,826 |
219,275/1,120,826 = 0.20 |
23,836,679 |
23,836,679 X 0.20 = 4,663,339 |
NGS 的标准化系数 = 选定样品的酵母unique reads数/其他样品的酵母unique reads数
附录 A:测试细胞对洋地黄皂苷的敏感性
在 CUT&RUN 实验步骤中,向缓冲液中添加洋地黄皂苷能促进细胞膜透化以及一抗和 pAG-MNase 酶进入细胞和胞核。因此,缓冲液中有足量的洋地黄皂苷对抗体和酶的成功结合以及靶向基因位点的消化至关重要。不同细胞系对毛地黄皂苷透化细胞显示不同的敏感性。虽然本实验步骤中建议的洋地黄皂苷量应足以对大多数细胞系或组织进行透化,但您可以使用本实验步骤测试您的特定细胞系或组织。我们发现,添加过量的洋地黄皂苷对本实验没有损害,因此无需生成浓度曲线。所以,进行快速测试以确定建议的洋地黄皂苷量是否适合你的细胞系就足够了。
在开始之前:
- 用一个 1.5 ml 试管收集 10,000 - 100,000 个细胞。对于组织,从 1 mg 组织中收集分解的细胞(第 I-C 部分步骤 1-13)。
- 在室温下以 600 x g 离心分离 3 分钟,然后移除液体。
注: 如果肉眼看不到细胞沉淀,我们建议在步骤 2 中对细胞悬浮液进行初始离心后,在不干扰细胞沉淀的情况下去除尽可能多的细胞培养基,并留下一些细胞 每个反应的培养基。然后在步骤 3 中,向细胞悬液中加入足够的 1X 洗涤缓冲液,使总体积达到 100 µl。
- 在 100 µl 洗涤缓冲液中重悬细胞沉淀。
- 将 2.5 µl 洋地黄皂苷溶液 #16359 添加到每个反应中,并在室温下孵育 10 分钟。
- 将 10 µl 细胞悬液与 10 µl 0.4% 台盼蓝染料混合。
- 使用血细胞计数器或细胞计数器计算染色细胞的数量和细胞总数。充分通透会导致 > 90% 的细胞被台盼蓝染色。
- 如果被台盼蓝染色的细胞不到 90%,则增加洋地黄皂苷缓冲液中添加的洋地黄皂苷溶液 #16359 的量,并重复步骤 1-5,直到 > 90% 的细胞被通透和染色。在第 I - IV 部分中采用上述洋地黄皂苷溶液 #16359 的量。
附录 B:对输入样品进行超声处理优化
建议对input DNA 样品进行超声处理,因为 DNA 纯化离心柱只能纯化片段化的基因组 DNA (< 10 kb)。此外,片段化的基因组 DNA (< 1 kb) 可在 NG-seq 分析中用作阴性对照。应优化超声处理,以便input DNA 的长度为 100-600 bp。
我们建议使用input样品进行 NG-seq,因为它能方便且无偏倚的呈现细胞基因组。虽然 IgG 样品也可在 NG-seq 中用作阴性对照,但由于存在非特异性结合,它可能会显示基因组特定区域出现富集。非片段化input DNA 可用于 qPCR 分析。但必须使用苯酚/氯仿提取后再行乙醇沉淀方法来纯化非片段化的 DNA。
在开始之前:
! 所有缓冲液的体积都应根据正在制备的input样品的数量按比例增加。
- 室温下取出并加热 DNA Extraction Buffer #42015,确保其完全融化成溶液。
- 每份 input 样品制备 2.1 ml 1X 洗涤缓冲液(210 µl 10X 洗涤缓冲液 #31415 + 1.89 ml 无核酸酶水#12931),并让其平衡到室温,以最大程度地减少细胞应激。无需向此洗涤缓冲液添加亚精胺或蛋白酶抑制剂混合物 #7012。
- 每份 input 样品制备 2 µl 蛋白酶 K #10012 + 0.5 µl RNAse A #7013,添加到 197.5 µl DNA Extraction Buffer #42015(每份 input 样品 200 µl)中。
- 在 1.5 ml 试管中,收集与您在 CUT&RUN 实验中用于输入的相同数量的细胞(5,000 到 100,000 细胞),用于测试的每个超声处理条件。对于组织,从用于 CUT&RUN 实验(第 I-C 部分步骤 1-13)输入的相同数量的组织中收集分离的细胞,用于测试的每个超声处理条件。
- 在室温下以 600 x g 离心分离 3 分钟,然后移除液体。
注: 如果在处理低细胞数(<100,000 个细胞)时肉眼看不到离心后的细胞沉淀,我们建议跳过下面的洗涤步骤 3-5。在步骤 2 中对细胞悬浮液进行初始离心后,在不干扰细胞沉淀的情况下去除尽可能多的细胞培养基,并在每个反应中留下一些细胞培养基。然后在步骤 6 中,将足够的 1X 洗涤缓冲液添加到细胞悬液中,以便在每个被测试的超声处理条件下达到 100 µl 的体积。
- 轻轻上下吹打,以在 1 ml 1X 洗涤缓冲液中重悬细胞沉淀物。
- 在室温下以 600 x g 离心分离 3 分钟,然后移除液体。
- 重复步骤 3 和 4一次来再次清洗细胞沉淀物。
- 加入 100 µl 的 1X 洗涤缓冲液,并通过轻轻上下吹打重悬细胞沉淀。
- 对于每个超声处理的条件,分装 100 µl 细胞悬液到一个新试管中。
注: 在步骤 9中,在 55 °C 下孵育样品,因此建议使用一个可封盖的 1.5 ml 管子,以减少孵育过程中出现蒸发。
- 向每份样品添加 200 µl DNA 提取缓冲液(+ 蛋白酶 K + RNAse A),并上下摇晃移液管来混合。
- 将试管放在 55°C 下 1 小时,保持摇晃混匀试管。
- 将试管放在冰上 5 分钟,让样品完全冷却。
- 设定一个以 15 秒脉冲声处理的周期数渐增的时程实验,以确定你的超声波仪的最佳声处理条件。确保在两次脉冲之间将样品放在冰上孵育 30 秒钟。
- 在 4°C 下用微量离心机以 18,500 x g 的离心力离心分离 10 分钟来使裂解物变澄清。将上清液转移到一根新的 2 ml 微量离心管。
- 按照第 VI 部分的说明,使用 DNA 纯化离心柱或使用苯酚/氯仿提取后再行乙醇沉淀方法来纯化 DNA 样品。
- 从离心柱中洗脱 DNA,或在 30 µl 1X TE 缓冲液或无核酸酶水 #12931 中重悬 DNA沉淀物。
- 通过电泳确定 DNA 片段大小。将 > 15 µl 样品上样到含 100 bp DNA 标准分子marker的 1% 琼脂糖凝胶上。建议使用无染料上样缓冲液(30% 甘油)来更好地观察凝胶上的 弥散DNA 。
- 选择能产生最佳 DNA 片段大小 (100-600 bp) 的超声处理条件,并用于第 V 部分步骤 4 中的 input 样品制备。如果未达到最佳声处理条件,则增加或减少超声波仪的功率设置或声处理周期数,并重复声处理时程实验。
附录 C:疑难排解指南
有关详细的疑难解答指南,请访问 https://learn-and-support/troubleshooting/cutandrun-troubleshooting-guide
发布时间 2019 年 11 月
修订时间 2024 年 3 月

